Bakteriophagen als vielversprechende Biokontrollwerkzeuge im Kampf gegen antibiotikaresistente Keime
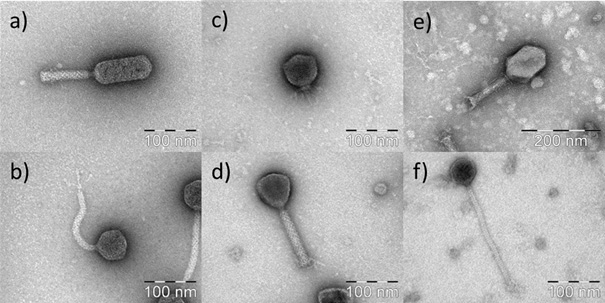
Der Einsatz von Bakteriophagen als Biokontrollwerkzeuge zur Erhöhung der Lebensmittelsicherheit wird zurzeit intensiv diskutiert. Auf der Suche nach einer wirksamen Methode zur Minimierung antibiotikaresistenter als auch humanpathogener Keime in Lebensmittel wurde im Rahmen des Projekts „Antibiotikaresistenzen“ eine Vielzahl verschiedener Bakteriophagen aus Umweltproben isoliert. Insgesamt wurden über 40 verschiedene Phagen anhand ihrer Morphologie, RAPD-Muster, Wirtsspektren und DNA-Restriktionsmuster im Detail charakterisiert. Die Phagen konnten morphologisch in die Familien der Siphoviridae, Myoviridae und Podoviridae (Abb.) eingeordnet werden. Für das Phagenscreening wurden verschiedene EHEC-Stämme, antibiotikaresistente E. coli-Isolate sowie Klebsiellen und Enterokokken verwendet. Zur genauen genetischen Charakterisierung werden die Genome ausgewählter Phagen zurzeit vollständig sequenziert.
Die Wirtsspektren der Phagen waren erstaunlich unterschiedlich. Einer der lytisch aktivsten Phagen wies ein breites Wirtsspektrum auf und infizierte alle getesteten EHEC-Stämme, während das Wirtsspektrum eines anderen Phagen auf E. coli O157:H- und O157:H7-Stämme eng begrenzt war. Wiederum ein anderer Phage zeigte eine speziesübergreifende lytische Aktivität sowohl für verschiedene antibiotikaresistente E. coli-Stämme, als auch für einen Salmonella-Stamm.
Aus Umweltproben konnten somit viele unterschiedliche Phagen für E. coli-, Klebsiella- und Vancomycin-resistente Enterococcus faecium bzw. faecalis-Bakterienstämme isoliert werden, die sich hinsichtlich ihrer lytischen Aktivität unterscheiden und die sich möglicherweise zur Biokontrolle von antibiotikaresistenten oder humanpathogenen Bakterien anbieten.